发布时间:2023-11-08 发布人:布平医疗
1. 前言
伴多层菊形团的胚胎性肿瘤(embryonal tumor with multilayered rosettes,ETMR)是一种罕见的儿童肿瘤,原属于中枢神经系统原始神经外胚层肿瘤(Central Nervous System-Primitive Neuroectodermal Tumor,CNS-PNET)的一种,但是CNS-PNET包含多个亚型及变异,以传统病理学对CNS-PNET分类不准确一直为临床病理诊断所困。最终因诊断标准之特异性有限以及误诊率较高,故世卫组织于2016年修订版中将CNS-PNET从CNS肿瘤分类中移除。相关后续研究通过DNA甲基化分型重新评估CNS-PNE参数,许多病变得以依据表观遗传学重新分为特定几类,其中已知者有多层菊形团的胚胎性肿瘤( embryonal tumor with multilayered rosettes,ETMR)、高级别胶质瘤(high-grade glioma,HGG)等[1]。现如今随着甲基化技术在中枢神经系统肿瘤当中越来越重要的检测地位,越来越多其他的中枢神经系统肿瘤也开始遵循基于DNA甲基化修饰的新分类标准,从而完善现有的病理诊断技术。
多层菊形团的胚胎性肿瘤 (ETMR)是罕见的胚胎性肿瘤。ETMR镜下以多层原始未分化细胞围绕血管腔呈放射状排列分布形成多层细胞菊形团为特征[2, 3]。多层菊形胚胎性肿瘤的病理特征包括多层的上皮样细胞,类似于胚胎期的组织结构,但与其他肿瘤或病变,如胚胎性脑肿瘤、肺内胚胎性肿瘤等通过传统病理手段进行诊断存在较大困难。因此2016年WHO中枢神经系统肿瘤分类(修订版第四版)根据FISH检测是否出现C19MC位点扩增分为C19MC变异型非特殊(not otherwise specified,NOS)型及髓上皮瘤[4]。基于大部分ETMR还具有其他特异性基因亚型[2],2021年WHO中枢神经系统肿瘤分类(第五版)增加DICER1变异型[5]。当然除C19MC/DICER1外还有例如LIN28A、MIR17HG、CTNNB1、肿瘤蛋白p53(tumor protein p53,TP53)等不同的基因亚型也会对患者的预后产生影响。
因此若仅依靠常规组织病理诊断手段或FISH的检测结果对ETMR进行诊断存在很大局限性,同时越来越多的证据表明甲基化谱、下一代测序等检测方法将对ETMR的提供更多更有力的诊断帮助[2, 6]。国际上甲基化分型已成为中枢神经系统肿瘤辅助诊断的重要技术手段之一,已被引入最新世界卫生组织(第五版)肿瘤分类中。甲基化分型已成功用于多种中枢神经系统肿瘤类型诊断当中,并且在许多情况下被证明比常规组织病理学更具特异性[2,4]。
2. 病史介绍
姓名:XXX 性别:男 年龄:4
肉眼检查:(右侧额顶占位)灰褐色脑组织一块,总体大小为9×7×3.5cm,切面灰白灰褐色,实性,质软。
病理诊断:(右侧额顶占位)高级别肿瘤,综合组织形态及免疫组化结果倾向为伴多层菊形团的胚胎性肿瘤,WHO4级,建议分子检测以明确分类分型。
免疫组化:瘤细胞SYN(+),NEUN(部分+),NSE部分(+),OLIG-2(少许+),INI-1(+,未缺失),PTEN(+),ATRX(+),MGMT约70%(+),P53(约60%+),CD34(-),GFAP(-),IDH-1(-),EMA(-),S-100(-),VIM(+),CK(-),Nestin(+),BRAF(-),H3K27M(-),H3K27ME3约40%(+),EGFR(约3%+),KI-67约80%(+)。

图1:患者多层菊形团的胚胎性肿HE染色图(扫描二维码查看HE染色实图)
甲基化分型器检测结果:临床考虑为胚胎性脑肿瘤,根据患者检测结果C19MC 拷贝数扩增,最终判定患者为:伴多层菊形团的胚胎性肿瘤,C19MC变异型。

图2:患者甲基化分型结果
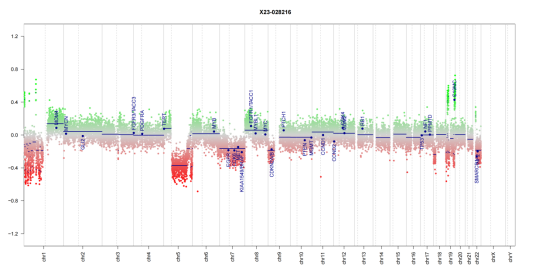
图3:拷贝数变异分析结果:C19MC 拷贝数扩增
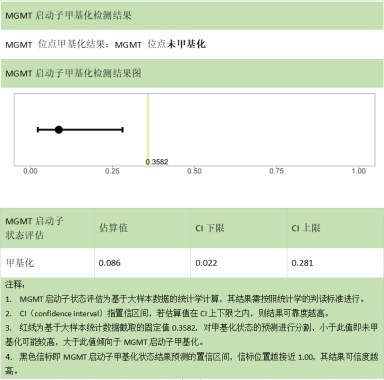
图4:MGMT启动子甲基化检测结果
3. 病例讨论
CNS-PNET依靠常规组织病理诊断标准之特异性有限以及误诊率较高,已经从2016年修订的WHO神经系统肿瘤分类中删除,但是后续通过DNA甲基化修饰的分析发现,CNS-PENT可以分为四类:含有伴多层菊形团的胚胎性肿瘤(ETMR)、高级别胶质瘤(HGG)、中枢神经母细胞瘤伴FOXR2基因激活(CNS NB-FOXR2)等。相关研究基于以上甲基化修饰分类,分析了307例患者的治疗方案以及预后,发现CNS NB-FOXR2患者接受手术后放化疗效果好,而ETMR的患者需要进一步根据DNA甲基化修饰的结果,寻找新的分子靶向治疗方案,进而提高此类患者的预后。
伴多层菊形团的胚胎性肿瘤(ETMR)通常发生在婴幼儿和幼儿期,主要发生在脑部,尤其是大脑半球。ETMR镜下以多层原始未分化细胞围绕血管腔呈放射状排列分布形成多层细胞菊形团为特征,依靠常规组织病理诊断标准之特异性有限以及误诊率较高,推荐采用其他分子生物学检测来进行诊断。
ETMR诊断涉及的生物标志物除了公认的C19MC/DICER1亚型标志物外,还存在其他具有很高的检测价值生物标志物。例如LIN28A是一种RNA结合蛋白,在ETMR中呈高表达,被认为是具有高度特异性和敏感性的ETMR标志物[7,8]。该蛋白具有增加癌细胞增殖、转移、血管生成、细胞死亡抗性和基因组不稳定性的功能[9]。除常见C19MC扩增和LIN28A高表达外[7,8,9,10],ETMR也存在其他较少见的标志物,包括MIR17HG扩增[11]、DICER突变[12]、CTNNB1的第3外显子突变[13]、肿瘤蛋白p53(tumor protein p53,TP53)纯合子或半合子突变等[14]。以上不同的不同的基因亚型仅依靠目前常规组织病理或现有FISH和NGS套餐也难以完全涵盖所有生物标志物,存在一定的限制,会对最终的病理诊断结果产生影响。相较而言,众多的研究表明几乎所有的肿瘤类型都具有特征性的甲基化谱,大多数CNS肿瘤类型都可以通过甲基化分型获得可靠的分类。相关的研究表明通过甲基化分型后可以准确区分ETMR与HGG和CNS NB-FOXR2患者的预后情况。本案例ETMR患者通过进行12.5版中枢神经系统肿瘤甲基化分型器检测C19MC 拷贝数扩增的结果。突破了常规组织病理诊断的局限性最终诊断为C19MC变异型伴多层菊形团的胚胎性肿瘤,也为后续的治疗给予了提供了可靠的指导意见。

图5:的拷贝数概况、性别比例、首次诊断的年龄分布、肿瘤位置、治疗及转归
总结:相较于常规组织病理诊断标准之特异性有限以及误诊率较高,甲基化分型技术将ETMR与CNS NB-FOXR2等中枢神经系统肿瘤区分出来,让患者得到更加针对性的治疗指导,进而提高此类患者的预后。ETMR拥有多个有价值的生物标志物的患者,仅依靠FISH或NGS检测结果对ETMR进行诊断还是存在一定的局限性。甲基化检测可以作为一种在CNS当中较为全面检测手段,在一定程度上让患者避免FISH和NGS的检测成本。后面通过甲基化谱分子诊断技术(CNS v12.5)通过一次检测所得到的相关参数结合医生的临床判断,最后准确地对该患者的肿瘤亚型进行准确地分类,也为患者的后续治疗提供了可靠的指导意义。
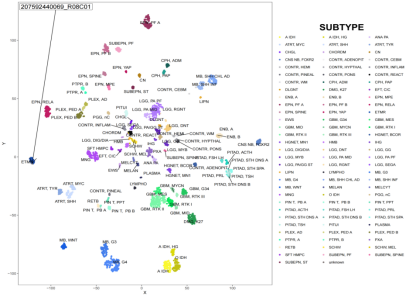
图6:基于甲基化分子特征的无监督聚类
近年来,甲基化分型已成为中枢神经系统肿瘤辅助诊断的重要技术手段之一,也越来越多的中枢神经系统肿瘤遵循基于DNA甲基化修饰的新分类标准。鉴于有关中枢神经系统肿瘤分型基础的研究取得了重大进展,WHO CNS5更加重视分子分型,细化了中枢神经系统肿瘤的分类,增加了有关大多数肿瘤的甲基化特征的内容[5,15],明确提出甲基化分型是肿瘤诊断的重要辅助手段[3,16]。布平医疗聚焦下一代病理诊断技术,针对中枢神经系统肿瘤推出甲基化谱分子诊断技术(CNS v12.5)帮助多数中枢神经系统(CNS)肿瘤根据DNA甲基化结果进行诊断,尤其特征并不显著的、罕见的肿瘤类型和亚型,帮助医生患者诊断分类,发掘新亚型肿瘤,提升对未认知肿瘤的识别。减少病理医生之间的主观诊断差异,帮助医生制定后续治疗方案。
4. 甲基化谱分子诊断技术(CNS v12.5)产品介绍
中枢神经系统肿瘤即使进行了多轮免疫组化与分子标志物检测,仍然难以进行精确的诊断或分型,严重影响了治疗方案的选择。研究表明几乎所有的肿瘤类型都具有特征性的甲基化谱,大多数CNS肿瘤类型都可以通过甲基化分型获得可靠的分类。甲基化分子分类器技术通过对大量样本进行甲基化谱特征的机器学习并建立相似度校正评分系统,最终实现对上百种中枢神经系统肿瘤亚型的人工智能精准分类并且有助于鉴定未知类型的新疾病亚型。
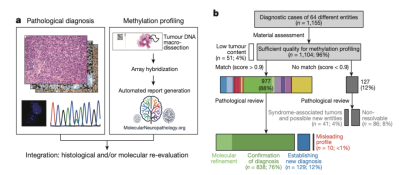
图7:一项发表在《Nature》上的多中心的双盲实验表明人为判定脑肿瘤的一致性和准确性较低。(绿色部分表示两组病理医生判断结果一致,其他则为判定结果不一致)
项目内容:
1.甲基化分型:主要检测目的,报告输出分型结果、相关解读及相似度得分。
2.全基因组染色体拷贝数变异:用于辅助鉴别不同的临床亚型,辅助肿瘤诊断。
3. MGMT启动子甲基化检测。

适用人群:

样本类型:

5.参考文献
[1] Von Hoff K, Haberler C, Schmitt-Hoffner F, et al. (2021). Therapeutic implications of improved molecular diagnostics for rare CNS embryonal tumor entities: results of an international, retrospective study. Neuro Oncol, 23(9):1597-1611.
[2] Sahm F, Schrimpf D, Stichel D, Jones DTW, Hielscher T, Schefzyk S, et al (2017) DNA methylation-based classification and grading system for meningioma: a multicentre, retrospective analysis. Lancet Oncol 18:682–694.
[3] Pickles JC, Stone TJ, Jacques TS (2020) Methylation-based algorithms for diagnosis: experience from neuro-oncology. J Pathol 250(5):510–517
[4] Capper D, Jones DTW, Sill M, Hovestadt V, Schrimpf D, Sturm D, et al (2018) DNA methylation-based classification of central nervous system tumours. Nature 555:469–474.
[5] Capper D, Jones DTW, Sill M, Hovestadt V, Schrimpf D, Sturm D et al (2018) DNA methylation-based classification of central nervous system tumours. Nature 555(7697):469–474
[6] Capper D, Jones DTW, Sill M, Hovestadt V, Schrimpf D, Sturm D et al (2018) DNA methylation-based classification of central nervous system tumours. Nature 555(7697):469–474
[7] Landgraf, P. , Rusu, M. , Sheridan, R. , Sewer, A. , & Tuschl, T. . (2007). A mammalian microrna expression atlas based on small rna library sequencing. Cell, 129(7), 1401-1414.
[8] Nguyen, P. N. N. , Choo, K. B. , Huang, C. J. , Sugii, S. , Cheong, S. K. , & Kamarul, T. . (2017). Mir-524-5p of the primate-specific c19mc mirna cluster targets tp53ipn1- and emt-associated genes to regulate cellular reprogramming. Stem Cell Research & Therapy, 8(1), 214.
[9] Lambo, S. , Hoff, K. V. , Korshunov, A. , Pfister, S. M. , & Kool, M. . (2020). Etmr: a tumor entity in its infancy. Springer Berlin Heidelberg(3).
[10] Bentwich IAvniel AKarov YAharonov RGilad SBarad OBarzilai AEinat PEinav UMeiri E. (2005). Identification of hundreds of conserved and nonconserved human micrornas. Nature Genetics, 37(7).
[11] Rao, S. , Rajeswarie, R. T. , Chickabasaviah Yasha, T. , Nandeesh, B. N. , Arivazhagan, A. , & Santosh, V. . (2017). Lin28a, a sensitive immunohistochemical marker for embryonal tumor with multilayered rosettes (etmr), is also positive in a subset of atypical teratoid/rhabdoid tumor (at/rt). Child\"s Nervous System.
[12] Rakheja, D. , Chen, K. S. , Liu, Y. , Shukla, A. A. , Schmid, V. , & Chang, T. C. , et al. (2014). Somatic mutations in drosha and dicer1 impair microrna biogenesis through distinct mechanisms in wilms tumours. Nature Communications, 2, 4802.
[13] Carsten, O. , Bueren, K. , Nicolas, U. , (2013). Treatment of young children with cns-primitive neuroectodermal tumors/pineoblastomas in the prospective multicenter trial hit 2000 using different chemotherapy regimens and radiotherapy. Neuro-oncology, 15(2), 224-234.
[14] Mong, E. F. , Yang, Y. , Akat, K. M. , Canfield, J. , & Totary-Jain, H. . (2020). Chromosome 19 microrna cluster enhances cell reprogramming by inhibiting epithelial-to-mesenchymal transition. Scientific Reports, 10(1).
[15] Priesterbach-Ackley LP, Boldt HB, Petersen JK, Bervoets N, Scheie D, Ulhøi BP et al (2020) Brain tumour diagnostics using a DNA methylation-based classifier as a diagnostic support tool. Neuropathol Appl Neurobiol 46(5):478–492
[16] Pickles JC, Fairchild AR, Stone TJ, Brownlee L, Merve A, Yasin SA et al (2020) DNA methylation-based profiling for paediatric CNS tumour diagnosis and treatment: a population-based study. Lancet Child Adolesc Health 4(2):121–130.